3.2. SciPy¶
A biblioteca SciPy disponibiliza muitas funcionalidades de computação científica implementadas de forma eficiente, incluindo integração numérica, interpolação, otimização, álgebra linear e Estatística. A documentação completa pode ser encontrada em docs.scipy.org/doc. Os módulos disponíveis no SciPy incluem:
cluster: Algoritmos de agrupamento hierárquicos e por quantização vetorial
constants: Constantes físicas e matemáticas
fftpack: Funções relacionadas à transformada rápida de Fourier
integrate: Funções para calcular integrais e equações diferenciais ordinárias
interpolate: Interpolação e splines de suavização
linalg: Álgebra linear
optimize: Otimização e funções para encontrar raízes
signal: Processamento de sinais
spatial: Algoritmos, estruturas de dados espaciais e distâncias
stats: Funções e distribuições estatísticas
3.2.1. Funções para integração¶
O módulo integrate oferece diversas funções para calcular integrais. A primeira delas é a função quad, que permite computar uma integral definida de uma variável:
from scipy.integrate import quad
x2 = lambda x: x**2
integral = quad(x2, 0, 2)
print('resultado: {}, erro: {}'.format(integral[0], integral[1]))
resultado: 2.666666666666667, erro: 2.960594732333751e-14
import numpy as np
def normal_fdp(x, mu, sigma_2):
return np.exp(-(x - mu) ** 2 / (2 * sigma_2)) / np.sqrt(2 * np.pi * sigma_2)
standard_fdp = lambda x: normal_fdp(x, 0, 1)
integral = quad(standard_fdp, -3, 3)
print('P(-3 < X < 3): {}, erro: {}'.format(integral[0], integral[1]))
P(-3 < X < 3): 0.9973002039367399, erro: 1.1072256503105314e-14
Para computar integrais duplas, triplas e sobre múltiplas variáveis, o módulo oferece respectivamente as funções dblquad, tplquad e nquad.
3.2.2. Funções para interpolação¶
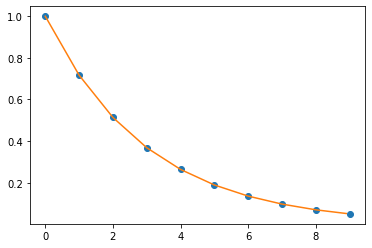
Para interpolar funções 1D, o ódulo interpolate possui a função interp1d. Essa função é chamada com dois vetores x e y e retorna uma função que pode ser chamda para usar interpolação e encontrar novos valores de y para novos valores de x.
import matplotlib.pyplot as plt
from scipy.interpolate import interp1d
x = np.arange(0, 10)
y = np.exp(-x / 3.0)
f = interp1d(x, y)
x_new = np.arange(0, 9, 0.1)
y_new = f(x_new)
plt.plot(x, y, 'o', x_new, y_new, '-')
plt.show()
3.2.3. Funções espaciais¶
O módulo spatial contém diversas funções para calcular distâncias, realizar transformações espaciais, encontrar envólucros convexos, etc. As masis de 20 funções de distâncias são encontradas no submódulo spatial.distance e incluem distâncias entre vetores e entre linhas de matrizes. A função pdist permite calcular as distâncias par-a-par entre as linhas de uma matriz X:
from scipy.spatial.distance import pdist
X = np.arange(12).reshape(4, 3)
print(pdist(X))
[ 5.19615242 10.39230485 15.58845727 5.19615242 10.39230485 5.19615242]
O retorno da função pdist é uma matriz condensada na forma de um vetor, para economizar espaço. A ideia é que a matriz de distâncias original tem diagonal 0 e valores espelhados. Para recuperar a matriz quadrada original, pode-se usar a função squareform:
from scipy.spatial.distance import squareform
print(squareform(pdist(X)))
[[ 0. 5.19615242 10.39230485 15.58845727]
[ 5.19615242 0. 5.19615242 10.39230485]
[10.39230485 5.19615242 0. 5.19615242]
[15.58845727 10.39230485 5.19615242 0. ]]
A função pdist permite escolher a métrica de distância utilizada, tendo a distância Euclidiana como padrão. Por exemplo, para se calcular a distância Euclidiana quadrática, pode-se fazer:
print(squareform(pdist(X, metric='sqeuclidean')))
[[ 0. 27. 108. 243.]
[ 27. 0. 27. 108.]
[108. 27. 0. 27.]
[243. 108. 27. 0.]]
Para calcular as distâncias entre as linhas de uma matriz \(A\) e as de uma matriz \(B\), usa-se a função cdist, que, assim como a função pdist, permite escolher a métrica.
from scipy.spatial.distance import cdist
A = np.arange(30).reshape(10, 3)
B = np.random.random((4, 3))
print(cdist(A, B, metric='cityblock'))
[[ 2.12698605 1.63700702 3.09875177 3.04020271]
[11.0632963 10.55103439 10.90092708 10.66965794]
[20.0632963 19.55103439 19.90092708 19.66965794]
[29.0632963 28.55103439 28.90092708 28.66965794]
[38.0632963 37.55103439 37.90092708 37.66965794]
[47.0632963 46.55103439 46.90092708 46.66965794]
[56.0632963 55.55103439 55.90092708 55.66965794]
[65.0632963 64.55103439 64.90092708 64.66965794]
[74.0632963 73.55103439 73.90092708 73.66965794]
[83.0632963 82.55103439 82.90092708 82.66965794]]
3.2.4. Funções de otimização¶
O módulo optimize oferece funções para minimização ou maximização de funções objetivo, com a possibilidade de forçar restrições. A principal função desse módulo é a minimize, que precisa receber como parâmetros uma função que será minimizada e uma solução inicial. Outros parâmetros incluem argumentos extras para a função minimizada, o método de minimização escolhido (padrão BFGS), possíveis restrições, uma função para calcular a Hessiana, um parâmetro e tolerância para critério de parada, entre outros.
from scipy.optimize import minimize
f = lambda x: x ** 2
x0 = 10
res = minimize(f, x0)
print(res.x)
[-4.14270251e-08]
x0 = 1
res = minimize(f, x0)
print(res.x)
[-7.45036449e-09]
x0 = 10
bounds = [(0, None)]
res = minimize(f, x0, bounds=bounds)
print(res.x)
[2.88291837e-15]
x0 = 1
bounds = [(0, None)]
res = minimize(f, x0, bounds=bounds)
print(res.x)
[0.]
A função minimize pode ser usada para estimar modelos. Por exemplo, vamos usá-la para estimar um modelo de regressão linear:
np.random.seed(42)
X = np.random.random(1000)
y = 3 * X + 2 + np.random.normal(size=1000)
betas = np.random.normal(size=2)
sq_err = lambda b, x, y: np.mean(((x*b[1] + b[0]) - y) ** 2)
res = minimize(sq_err, betas, args=(X, y))
print(res.x)
[2.17478032 2.84521493]
3.2.5. Funções estatísticas¶
O módulo stats contém uma grande quantidade de distribuições de probabilidade (124 atualmente), e uma biblioteca em constante crescimento de funções estatísticas, incluindo estatística descritiva, distribuições de frequência, correlações, testes, transformações, distâncias, estatística circular, etc. Também há duas classes que facilitam a criação de distribuições customizadas: rv_continuous, para distribuições contínuas, e rv_discrete, para discretas.
3.2.5.1. Usando distribuições¶
Nesta Seção, tomaremos como exemplo a distribuição normal, representada pela classe scipy.stats.norm, que herda todos métodos genéricos da classe rv_continuous. A classe norm representa a forma normal padrão, i.e. sua função densidade de probabilidade (fdp) para um número real \(x\) é: $\(f(x) = \frac{e^{-x^{2}/2}}{\sqrt{2\pi}}.\)$ Para importar a ditribuição normal, fazemos:
from scipy.stats import norm
Para calcular alguns momentos:
mean, var, skew, kurt = norm.stats(moments='mvsk')
print('média: {}, variância: {}, assimetria: {}, curtose: {}'.format(mean, var, skew, kurt))
média: 0.0, variância: 1.0, assimetria: 0.0, curtose: 0.0
Como dito acima, essa classe implementa a distribuição normal padrão. Para deslocar ou mudar a escala da distribuição, i.e. informar média e desvio-padrão, pode-se infomar os parâmetros loc e scale, respectivamente. Exemplo:
mean, var = norm.stats(loc=2, scale=0.5, moments='mv')
print('média: {}, variância: {}'.format(mean, var, skew, kurt))
média: 2.0, variância: 0.25
Para calcular a fdp para um número real \(x\), fazemos:
x = 1
print(norm.pdf(x, loc=2, scale=0.5))
0.10798193302637613
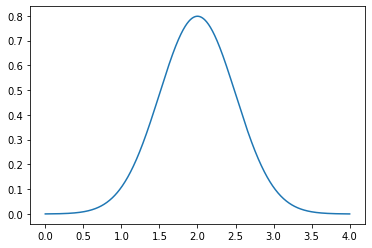
A função pdf também pode ser usada para calcular a fdp para cada elemento de um array:
import numpy as np
import matplotlib.pyplot as plt
x = np.linspace(0, 4, 1000)
plt.plot(x, norm.pdf(x, loc=2, scale=0.5))
plt.show()
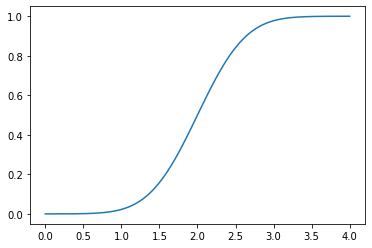
A função de distribuição acumulada pode ser calculada de maneira muito similar, usando a função cdf:
plt.plot(x, norm.cdf(x, loc=2, scale=0.5))
plt.show()
Para construir o intervalo que contém \(95\%\) da distribuição, faz-se:
print(norm.interval(0.95, loc=2, scale=0.5))
(1.020018007729973, 2.979981992270027)
Para construir o intervalo de confiança de \(95\%\) para a média de \(100\) observações de uma normal \(X \sim N(2, 0.25)\), podemos usar a função interval novamente:
print(norm.interval(0.95, loc=2, scale=0.5/100))
(1.9902001800772997, 2.0097998199227)
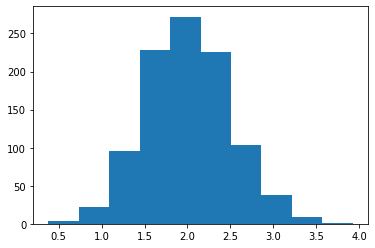
Para retirar amostras de uma variável \(X \sim N(2, 0.25)\), usamos a função rvs (de random variable sample):
np.random.seed(42)
samples = norm.rvs(loc=2, scale=0.5, size=1000)
plt.hist(samples)
plt.show()
Para obter os percentis de uma variável \(X \sim N(2, 0.25)\) usamos a função ppf (percent point function):
q1 = norm.ppf(0.25, loc=2, scale=0.5)
q3 = norm.ppf(0.75, loc=2, scale=0.5)
print('primeiro quartil: {}, terceiro quartil: {}'.format(q1, q3))
primeiro quartil: 1.6627551249019592, terceiro quartil: 2.337244875098041
Note que, ao longo desses exemplos, foi necessário informar a média (loc) e o desvio padrão (scale) repetidamente. Para evitar essa repetição de código, é possível criar um objeto norm que armazena a média e o desvio padrão para posterior uso nos demais métodos. Para isso, basta chamar norm em forma de função (construtor).
rv = norm(loc=2, scale=0.5)
samples = rv.rvs(size=1000)
plt.hist(samples)
plt.show()
x = np.linspace(0, 4, 1000)
plt.plot(x, rv.pdf(x))
plt.show()
Ao fazer isso, os métodos não aceitarão mais os parâmetros loc e scale:
plt.plot(x, rv.pdf(x, loc=2, scale=0.5))
plt.show()
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
<ipython-input-25-c0d6c5024495> in <module>
----> 1 plt.plot(x, rv.pdf(x, loc=2, scale=0.5))
2 plt.show()
TypeError: pdf() got an unexpected keyword argument 'loc'
Outras funções úteis incluem expect e fit. A função expect calcula o valor esperado de uma função com respeito à distribuição. Exemplo:
def square(x):
return x ** 2
expected_x_square = rv.expect(square)
square_expected_x = rv.mean() ** 2
var_calc = expected_x_square - square_expected_x
print('variância 1: {}, variância 2: {}'.format(var_calc, rv.var()))
variância 1: 0.2500000000000009, variância 2: 0.25
A função fit ajusta uma distribuição ao array de dados informado. No caso da distribuição normal, isso significa calcular a média e o desvio padrão dos dados. Exemplo:
np.random.seed(42)
x = norm.rvs(loc=1, scale=3, size=1000)
mean, std = norm.fit(x)
print('média: {}, desvio padrão: {}'.format(mean, std))
média: 1.0579961674669767, desvio padrão: 2.9361786232420632
Apesar de termos usado a distribuição normal como exemplo, todas as funcionalidades que apresentamos também se aplicam às outras distribuições contínuas e discretas, com uma diferença: nas distribuições discretas, a função pdf dá lugar à função pmf (função massa de probabilidade). No entando, seu uso é igual.
3.2.5.2. Testes de hipótese¶
O módulo stats atualmente oferece 32 funções para realizar diferentes testes de hipótese, incluindo testes paramétricos e não-paraétricos para média, mediana e variância e testes de bondade de ajuste e de normalidade. Começaremos cobrindo o teste t de Student bicaudal para a média de uma coleção de observações, i.e. \(H_0\text{:}~ \bar{x} = \mu_0\) e \(H_1\text{:}~ \bar{x} \neq \mu_0\). Esse teste pode ser realizado pela função ttest_1samp, que recebe como parâmetros um array e uma média populacional:
from scipy.stats import ttest_1samp
np.random.seed(42)
x = norm.rvs(loc=5, scale=10, size=1000)
print(
'Não rejeita a hipótese nula: {}'.format(ttest_1samp(x, 5))
)
print(
'Rejeita a hipótese nula: {}'.format(ttest_1samp(x, 0))
)
Não rejeita a hipótese nula: Ttest_1sampResult(statistic=0.6243089585093202, pvalue=0.5325672436623019)
Rejeita a hipótese nula: Ttest_1sampResult(statistic=16.77129726247285, pvalue=8.210083235562808e-56)
Os testes t para duas amostras podem ser realizados para amostras independentes ou pareadas por meio das funções ttest_ind e ttest_rel, respectivamente. A função ttest_ind recebe dois arrays e realiza um teste bicaudal para igualdade das duas médias. Exemplo:
from scipy.stats import ttest_ind
np.random.seed(42)
a = norm.rvs(loc=5, scale=10, size=50)
b = norm.rvs(loc=5, scale=10, size=30)
print(
'Não rejeita a hipótese nula: {}'.format(ttest_ind(a, b))
)
Não rejeita a hipótese nula: Ttest_indResult(statistic=-1.2296429533938804, pvalue=0.22252704557384642)
A função ttest_ind assume por padrão que as duas populações têm a mesma variância. Caso deseje-se assumir variâncias diferentes, pode-se atribuir o valor False ao parâmetro equal_var:
np.random.seed(42)
a = norm.rvs(loc=5, scale=10, size=50)
b = norm.rvs(loc=0, scale=1, size=30)
print(
'Rejeita a hipótese nula: {}'.format(ttest_ind(a, b, equal_var=False))
)
Rejeita a hipótese nula: Ttest_indResult(statistic=2.0257732965417956, pvalue=0.048054285617342485)
Caso apenas os valores das médias, desvios e tamanhos amostrais estejam disponíveis (ao invés das amostras em si), pode-se usar a função ttest_ind_from_stats:
from scipy.stats import ttest_ind_from_stats
mean1, std1, nobs1 = 0, 1, 50
mean2, std2, nobs2 = -1, 1, 15
print(
'Rejeita a hipótese nula: {}'.format(
ttest_ind_from_stats(
mean1, std1, nobs1, mean2, std2, nobs2, equal_var=False
)
)
)
Rejeita a hipótese nula: Ttest_indResult(statistic=3.396831102433787, pvalue=0.0024703403870314903)
Para realizar testes t bicaudais para a igualdade de médias com amostras pareadas, o módulo stats disponibiliza a função ttest_rel:
from scipy.stats import ttest_rel
np.random.seed(42)
a = norm.rvs(loc=5, scale=10, size=50)
b = norm.rvs(loc=0, scale=1, size=50) # mesmo tamanho
print(
'Rejeita a hipótese nula: {}'.format(ttest_rel(a, b))
)
Rejeita a hipótese nula: Ttest_relResult(statistic=2.07798026408817, pvalue=0.042971567080910246)
O teste dos postos sinalizados de Wilcoxon é comumente tratado como uma versão não-paramétrica do teste t para amostras pareadas. Ele testa se a distribuição das diferenças entre as duas amostras é simétrica e centrada em zero. No módulo stats, esse teste é realizado pela função wilcoxon e inclui um parâmetro para escolha da cauda do teste, com padrão bicaudal (two-sided):
from scipy.stats import wilcoxon
np.random.seed(42)
a = norm.rvs(loc=5, scale=10, size=50)
b = norm.rvs(loc=6, scale=1, size=50)
print(
'Rejeita a hipótese nula: {}'.format(wilcoxon(a, b))
)
print(
'Rejeita a hipótese nula: {}'.format(
wilcoxon(a, b, alternative='two-sided') # mesmo resultado
)
)
print(
'Não rejeita a hipótese nula: {}'.format(
wilcoxon(a, b, alternative='greater')
)
)
print(
'Rejeita a hipótese nula: {}'.format(
wilcoxon(a, b, alternative='less')
)
)
Rejeita a hipótese nula: WilcoxonResult(statistic=388.0, pvalue=0.016018532187551015)
Rejeita a hipótese nula: WilcoxonResult(statistic=388.0, pvalue=0.016018532187551015)
Não rejeita a hipótese nula: WilcoxonResult(statistic=388.0, pvalue=0.9919907339062245)
Rejeita a hipótese nula: WilcoxonResult(statistic=388.0, pvalue=0.008009266093775507)
Para realizar o teste de Kolmogorov-Smirnov (KS) de bondade de ajuste é simples, basta informar uma amostra e o nome de uma distribuição disponível no módulo stats à função kstest:
from scipy.stats import kstest
np.random.seed(42)
a = norm.rvs(size=50)
print(
'Não rejeita a hipótese nula: {}'.format(
kstest(a, 'norm')
)
)
a = norm.rvs(loc=5, scale=10, size=50)
print(
'Rejeita a hipótese nula: {}'.format(
kstest(a, 'norm')
)
)
Não rejeita a hipótese nula: KstestResult(statistic=0.13357070563896067, pvalue=0.3074515771790649)
Rejeita a hipótese nula: KstestResult(statistic=0.6375899529148361, pvalue=2.827049477141131e-20)
Note que nos exemplos acima, a hipótese nula foi rejeitada quando os dados não foram sorteados de uma normal padrão. Isso porque, como vimos acima, no SciPy, norm é a distribuição padrão. No entanto, ao invés de passar o nome da distribuição como parâmetro, podemos passar a função cdf de uma distribuição normal qualquer. Exemplo
a = norm.rvs(loc=5, scale=10, size=50)
normal = norm(loc=5, scale=10)
print(
'Não rejeita a hipótese nula: {}'.format(
kstest(a, normal.cdf)
)
)
Não rejeita a hipótese nula: KstestResult(statistic=0.08326080724304702, pvalue=0.878828192065557)
Para testar a hipótese de que duas amostras foram extraídas da mesma distribuição contínua, podemos usar o teste KS para duas amostras:
from scipy.stats import ks_2samp
a = norm.rvs(loc=5, scale=10, size=50)
b = norm.rvs(loc=5, scale=10, size=50)
print(
'Não rejeita a hipótese nula: {}'.format(
ks_2samp(a, b)
)
)
b = norm.rvs(size=50)
print(
'Rejeita a hipótese nula: {}'.format(
ks_2samp(a, b)
)
)
Não rejeita a hipótese nula: Ks_2sampResult(statistic=0.18, pvalue=0.3959398631708505)
Rejeita a hipótese nula: Ks_2sampResult(statistic=0.68, pvalue=2.6679407140599687e-11)
